Metacherchant软件揭示了抗生素抗性的新原因
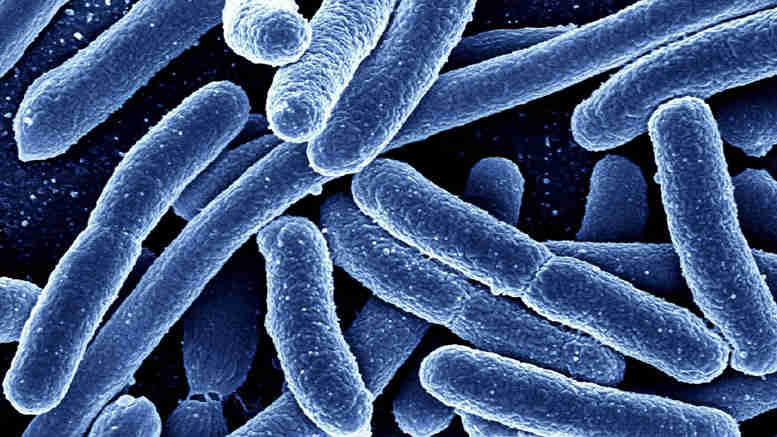
ITMO大学和物理化学医学中心的科学家开发了一种能够跟踪肠道微生物肿瘤中抗生素抗性基因传播的算法,并揭示了不同细菌物种之间的抗性基因的额外证据。该方法不仅可以促进有效治疗方案的发展,还可以抑制超前的传播。
近年来,抗生素抗性的传播成为全球医疗问题。由于抗生素在医学和农业中过度使用,肠道微生物群在其DNA或偏塞组中积聚抗生素抗性基因。一方面,这些基因有助于正常的植物生存。然而,另一方面,最近的研究表明,肠道微生物会能够与病原体共享抗性基因,从而使它们抵抗可用的疗法。在这种光明中,研究抵抗基因传播的研究变得尤为重要。
来自ITMO大学的程序员与物理和化学医学研究中心的同事制定了一种称为Metacherchant的算法,可以探索耐药基因环境,并了解它如何根据细菌种类而变化。“我们创建了一种工具,使科学家能够仔细看看两种或更多种微生物群样品中基因周围环境之间的差异。我们可以分析来自不同人群或在不同时期收集的微生物群样本,例如抗生素治疗前后的不同时间,“ITMO大学计算机技术部副教授Vladimir Ulyantsev说。“基于所获得的数据,我们可以建议特定的抗性基因如何从一种微生物物种传播到另一个微生物物种。”
对抗生素抗性基因环境的研究主要是设计有效抗微生物治疗方案的重要性。“使用MetacherChant,我们可以分析Microbiota如何为特定抗生素类的抗性产生贡献。期待着,可以预测抗生素,抗性最有可能在病原体中传播。另一方面,我们可以找到具有低抵抗风险的药物。反过来,这将有助于我们调整和调整特定的疗法。这是未来几年的问题,“Evgenii Olekhnovich,Levenii Olekhnovich,Lead Author And Measher Colity医学中心研究员说。
该算法的潜在应用不限于肠道微生物A基因分析,因为该程序也可用于研究来自土壤,水或污水的基因组样品。“我们可以评估单一细菌群落中抗性的差异,例如肠道微生物群,以及不同的社区之间。例如,这使我们能够识别通过环境传播的抗生素抗性的全局途径,“Evgenii Olekhnovich说。“阻力问题很复杂,需要一种复杂的方法,我们的工具可以真正有用。”
出版物:Evgenii I Olekhnovich等,等,“Metacherchant:分析肠道微生物的抗生素抗性基因的基因组背景,”生物信息学“,2017年; DOI:10.1093 / Bioinformatics / BTX681
-
生物学家揭示有丝分裂后细胞如何重建
2022-01-21 -
研究人员识别出于发育基因的可逆性“主切换”
2022-01-21 -
研究人员查明了乳腺癌风险背后的分子机制
2022-01-21 -
古代微生物在宇宙中提供了强大的生命证据
2022-01-21 -
科学家鉴定出与黑色素瘤进展相关的基因
2022-01-20 -
生物学家揭示了微生物在火星上可以生存多长时间
2022-01-20 -
遗传学家显示温度变化使基因编辑更有效
2022-01-19 -
Neu5GC糖与人类的自发肿瘤的出现有关
2022-01-18 -
肠道微生物可以消耗足够的关键营养素来引起缺陷
2022-01-18 -
生物学家对克罗地亚温迪贾洞穴的一个新的尼安德特人基因组进行测序
2022-01-18 -
发现有助于研究人员了解生活如何在其他行星上生存
2022-01-18 -
关于时钟基因的新洞察力可能会持有人脑进化的答案
2022-01-17 -
合成蛋白组装体封装自身的遗传物质并进化
2022-01-16 -
新的纳米药物抑制胰腺癌的进展
2022-01-16 -
新研究显示蜱蛋白IAFGP有助于抗生素作战MRSA
2022-01-12